** 이 포스트에서는 mRNA의 단백질으로의 번역 과정을 설명하기 위한 배경 지식들을 정리합니다.
↓ RNA 번역에 대한 내용은 아래의 포스트를 참고해주시면 됩니다.
[분자생물학] RNA 번역 과정과 특성 (개시, 신장, 종결) (미완성)
개시 : mRNA에 리보솜 소단위체가 결합하고, 개시코돈 AUG에 메티오닌(Met)을 가진 tRNA가 P 자리에 붙고, 그 다음 대단위체가 붙습니다. 신장 : 다음 아미노산을 가진 tRNA가 A 자리에 붙고, 아미노
restudycafe.tistory.com
mRNA 코돈 해독 실험
시험관 내 조건(in vitro)에서 RNA 서열에 따라 합성되는 단백질을 분석하여 RNA를 해독한 실험입니다.
인공 mRNA와 세균 추출물을 이용해서 원핵 생물의 유전자 발현 체계와 같고, 개시/종결 코돈이 필요없으며,
폴리뉴클레오티드 인산화효소를 이용하여 주형 가닥이 없어도 단위체를 연결할 수 있는 조건에서 진행하였습니다.
가장 먼저, 서열이 UUU로만 이루어진 mRNA를 이용하여 페닐알라닌으로만 이루어진 폴리펩티드가 합성된 것을 통해,
UUU 코돈에 해당하는 아미노산은 페닐알라닌임을 알아냈습니다. (AAA, CCC도 마찬가지로 실험)
점돌연변이가 발생했을 때 아미노산 3개에만 돌연변이가 일어난 것을 통해 중첩 해석이 일어남을 알아냈습니다.
(비중첩 해석이 일어났다면 뒤의 아미노산들이 다 밀려서 해석되어야 합니다.)
ATP와 CTP를 5:1의 비율로 넣은 뒤 RNA 합성 및 번역을 통해 얻어낸 아미노산 비율을 통해 일부를 해독하였습니다.
(다른 뉴클레오티드를 다른 비율로도 투입하여 다른 조합의 코돈들도 해석함)
마지막으로 (UG)n, (UUC)n과 같은 반복서열을 이용하여 번역되는 아미노산들을 분석하였습니다.
예를 들어 (GUU)n mRNA에 여러 tRNA들을 통과시키면 val-tRNA만 결합하여 여과지에 남습니다.
코돈 / 안티코돈
코돈은 mRNA의 3개의 뉴클레오티드로 구성된 서열로, 4^3 = 64가지가 존재하며, 하나의 아미노산으로 번역됩니다.
+ 개시 코돈은 AUG, 종결 코돈은 UAA, UAG, UGA입니다.
+ 모든 생명체는 동일한 코돈표를 사용하고 있습니다. (원핵 생물의 개시 코돈만 빨리 개시하는 fMet를 사용)
안티코돈은 코돈과 상보적인 tRNA의 특정 염기서열로, 45가지만 존재합니다.
그럼에도 코돈 한 종류는 안티코돈 여러 종류와 결합하는데, 안티코돈의 변형된 하나의 염기인 이노신(I)이 A, U, C와 상보적인 결합이 가능하기 때문에, 하나의 tRNA가 두 가지 이상의 코돈을 인식할 수 있는 것입니다.
따라서 안티코돈의 1번 염기(코돈의 3번 염기)가 엄격하지 않고, (워블 법칙)
리보솜 방출이 쉽고, 뉴클레오티드가 바뀌어도 같은 아미노산이 연결되기도 합니다. (동의 돌연변이)
충전 효소 : tRNA와 아미노산의 결합
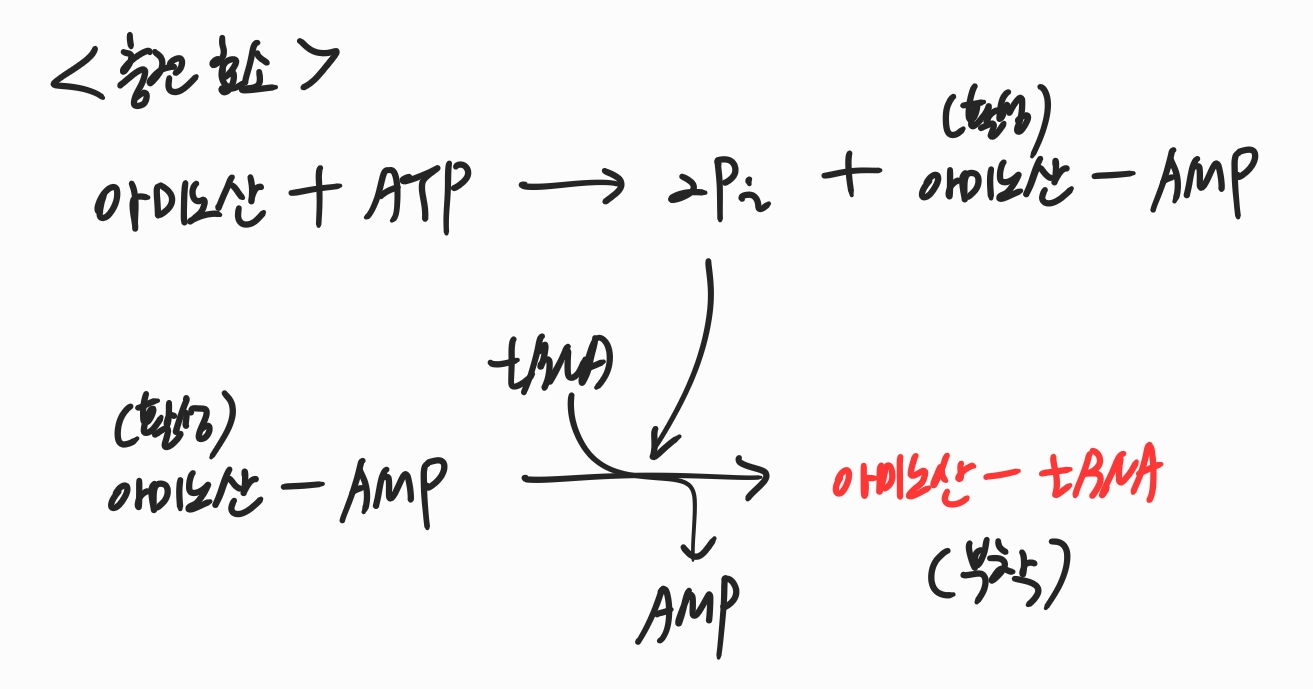
충전 효소는 ATP(또는 AMP)를 사용하여 아미노산을 tRNA에 부착시킵니다.
(단백질을 합성하는 리보솜으로 tRNA가 아미노산을 옮겨줘야 하는데, 그 준비과정인 것입니다.)
이 때 tRNA의 3' 말단(CCA)에 있는 A에 탈수 축합 반응을 일으켜 에스테르 결합을 형성합니다.
충전 효소는 아미노산별로 20가지 종류가 있으며, 교정 수선의 기능도 가지고 있습니다.
Cys-tRNA의 아미노산을 변형시켜 Ala-tRNA로 만들었음에도 mRNA 서열과 관계없이 Ala로 연결되는 실험을 통해,
(tRNA에 부착되는 아미노산의 종류와 관계없이) 코돈과 안티코돈의 상호작용으로 합성이 이루어짐을 알 수 있습니다.
리보솜
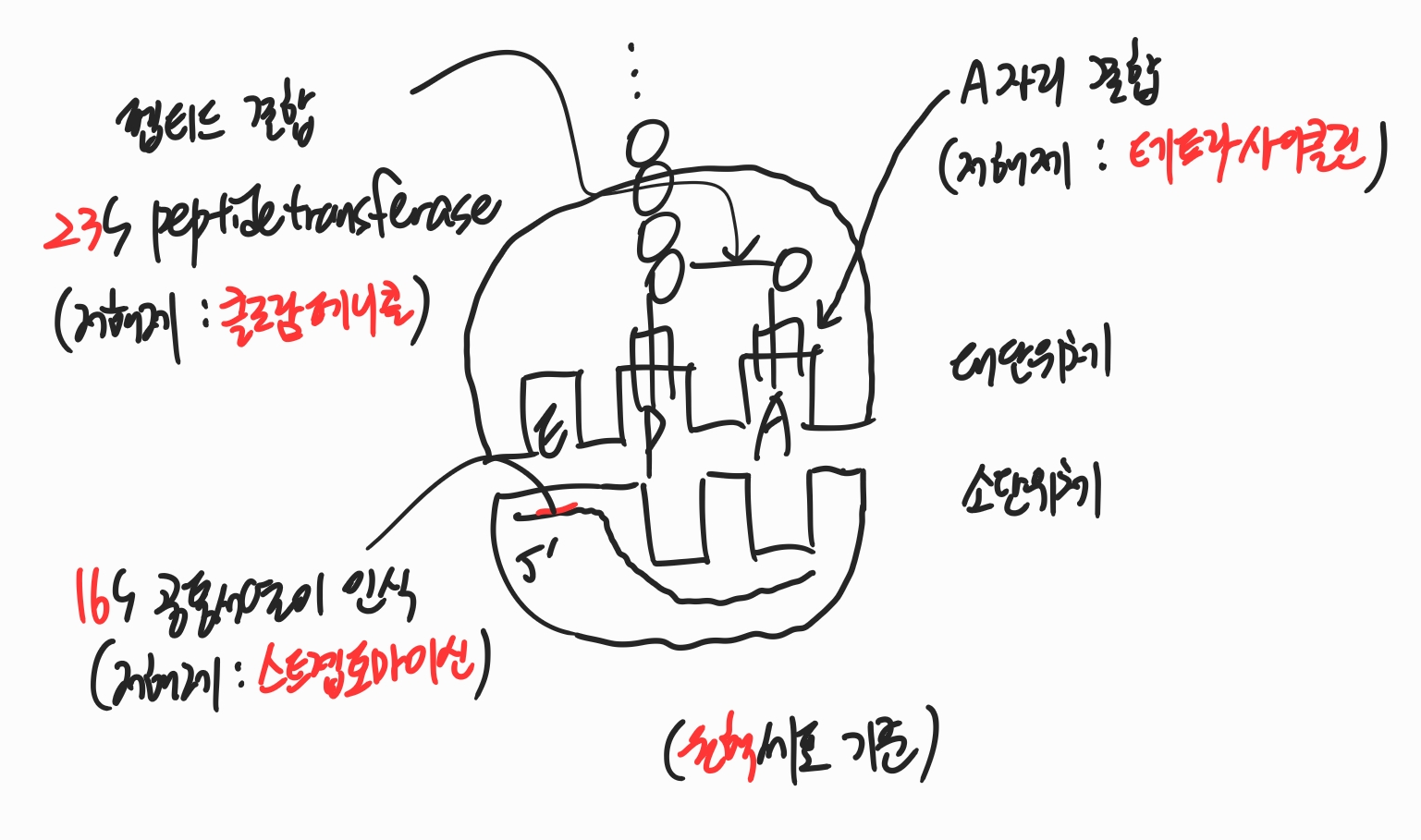
리보솜은 아미노산을 연결하여 단백질을 합성하는 세포소기관으로, rRNA + 단백질로 구성되며 E, P, A 자리가 있습니다.
원핵 세포의 리보솜은 50S 대단위체(5S + 23S)와 30S 소단위체(16S)로 이루어져 있으며,
(+ 16S의 공통서열 인식 저해 스트렙토마이신, A 자리 결합 저해 테트라사이클린, 23S 펩티드결합 저해 클로람페니콜)
진핵 세포의 리보솜은 60S 대단위체(5S + 5.8S + 28S)와 40S 소단위체(18S)로 이루어져 있습니다.
IRES (= UTR)
IRES(Internal Ribosome Entry Site)는 리보솜에 결합하는 선형(진핵 생물)의 mRNA 5' 말단에 있는 공통 서열입니다.
원핵 생물의 폴리시스트론 mRNA에 IRES를 추가해주면, 진핵 생물에서도 번역 및 발현시킬 수 있습니다.
** 원핵 생물에서는 번역 개시 과정에서 16S rRNA가 샤인-달가노 서열을 인식합니다.
mRNA 정제
Chloroform을 넣고 원심분리 시켜주면 상층액에 RNA만 분리됩니다.
상층액만 분리하여 Isopropanol을 넣고 원심분리하면 RNA만 침전됩니다.
침전물만 분리하여 70% 에탄올을 넣고 원심분리하면 RNA 응집체가 염세척됩니다.
'자연과학(PEET) > 생명과학' 카테고리의 다른 글
| [분자생물학] 번역 이후 단백질 이동/가공 과정 (0) | 2021.04.01 |
|---|---|
| [분자생물학] RNA 번역 과정과 특성 (개시, 신장, 종결) + 폴리리보솜과 번역 항생제 (0) | 2021.03.31 |
| [분자생물학] RNA 가공 과정과 특성 (mRNA capping/splicing/tailing, tRNA, rRNA) (0) | 2021.03.29 |
| [분자생물학] DNA 전사 과정과 특성 (개시, 신장, 종결) (0) | 2021.03.28 |
| [분자생물학] DNA 복제 과정과 특성 (개시, 신장, 종결) (0) | 2021.03.26 |